Взаимодействие miRNA с mRNA генов участвующих в метаболизме липидов
Конференция: XIX Международная научно-практическая конференция «Научный форум: медицина, биология и химия»
Секция: Математическая биология, биоинформатика

XIX Международная научно-практическая конференция «Научный форум: медицина, биология и химия»
Взаимодействие miRNA с mRNA генов участвующих в метаболизме липидов
Interaction of miRNA with mRNA genes involved in lipid metabolism
Aigul Akimniyazova
PhD-student of al-Farabi Kazakh National University, Kazakhstan, Almaty
Bakytzhamal Gabdulkhayeva
Candidate of biological sciences, associate ProfessorinPavlodar State Pedagogical University, Kazakhstan, Pavlodar
Zhadra Kdyrbayeva
Master student of al-Farabi Kazakh National University, Kazakhstan, Almaty
Аннотация. В статье с помощью биоинформатических подходов установлены количественные характеристики взаимодействия miRNA с mRNA генов участвующих в метаболизме липидов. Выявлены особенности расположения сайтов связывания miRNA в 5'UTR, CDS и 3'UTR mRNA генов FAS, FDPS, HSL, INSIG1, LDLR, SCAP и SREBP.
Abstract. Using bioinformatics approaches quantitative characteristics of miRNA interaction with mRNA of genes involved in lipid metabolism are established in the article. There are revealed the features of the arrangement of miRNA binding sites in 5'UTR, CDS and 3'UTR mRNA of FAS, FDPS, HSL, INSIG1, LDLR, SCAP and SREBP genes.
Ключевые слова: miRNA; mRNA; ген; метаболизм; липиды.
Keywords: miRNA; mRNA; gene; metabolism; lipids.
Введение. Метаболизм липидов является важной составляющей обмена веществ в организме и одно из направлений его изучения заключается в регуляции экспрессии генов участвующих в этих процессах. miRNA оказывают влияние на многие физиологические процессы путем регуляции экспрессии генов на стадии трансляции mRNA [1]. Нарушение метаболизма липидов вызывает ряд заболеваний и поэтому важно выяснить могут ли miRNA участвовать в развитии этих патологий. Описано влияние miRNA на метаболизм липидов, однако не установлено на какие гены мишени могут эти miRNA оказывать прямое влияние [2]. Нами были выбраны десять генов человека, которые участвуют в метаболизме липидов и связаны в генные сети [3].
Материалы и методы исследования. Нуклеотидные последовательности генов DGAT, FAS, FDPS, HMGCL, HSL, INSIG1, LDLR, PAIP1, SCAP и SREBP участвующих в метаболизме липидов, были взяты из GenBank (http://www.ncbi.nlm.nih.gov). Сайты связывания miRNA в 5'-нетранслируемой области (5'UTR), белок-кодирующей области (CDS) и 3'-нетранслируемой области (3'UTR) нескольких генов были предсказаны с помощью программы MirTarget [4]. Эта программа определяет следующие особенности связывания: a) начало сайта связывания miRNA с mRNA; б) расположение сайтов связывания в 5'UTR, CDS и 3'UTR mRNA; в) свободную энергию гибридизации (∆G, kJ/mole); и г) схемы взаимодействия нуклеотидов miRNA с mRNA генов-мишеней. Для каждого сайта рассчитывали отношение ΔG/ΔGm (%), где ΔGm равно свободной энергии связывания miRNA с полностью комплементарной нуклеотидной последовательностью. Сайты связывания miRNA с mRNA отбирали соотношением ΔG/ΔGm равным 90% и более. Позиция сайтов связывания указана от первого нуклеотида 5'UTRmRNA. Программа MirTarget учитывает взаимодействия нуклеотидов miRNA с mRNA генов-мишеней не только между аденином (А) и урацилом (U), гуанином (G) и цитозином (C), но и между A и C, G и U посредством одной водородной связи. Расстояние между A и C равно расстоянию между нуклеотидами G и C, A и U,G и U. [5-6]. Число водородных связей во взаимодействиях G-C, A-U, G-U и A-C равно 3, 2, 1 и 1 соответственно.
Результаты и обсуждение. 2567 miRNA могли влиять на гены FAS, FDPS, HSL, INSIG1, LDLR, SCAP и SREBP. Гены DGAT, HMGCL и PAIP1 не были мишенями изученных miRNA, либо в меньшей свободной энергией взаимодействовали с ними. Количественные характеристики взаимодействия miRNA с mRNA этих генов приведены в таблице 1. Полученные данные показывают, что изученные гены в разной степени зависели от miRNA. Ген FAS являлся мишенью четырех miRNA, ген HSL – пяти miRNA, ген SCAP – шести miRNA, гены LDLR и SREBP – восьми miRNA.
Распределение сайтов связывания miRNA в mRNA было неравномерным. Большая часть miRNA имели сайты связывания в разных участках mRNA и могли связываться независимо друг от друга. Однако некоторые miRNA имели сайты связывания с перекрывающимися нуклеотидными последовательностями. Такое расположение сайтов связывания miRNA может иметь несколько причин.
Сайты связывания miR-1273a и miR-1273g-3p в 5'UTR mRNA гена FAS перекрывались на пять нуклеотидов, что делает невозможным одновременное связывание этих miRNA. Преимущество в связывании с mRNA имеет miR-1273a, поскольку она имеет большую свободную энергию взаимодействия по сравнению с miR-1273g-3p (Таблица 1).
Такое преимущество будет иметь место при равных концентрациях этих miRNA.
Таблица 1.
Характеристики взаимодействия miRNA с mRNA генов участвующих в метаболизме липидов
|
Ген |
miRNA |
Начало сайта, нт |
Участок mRNA |
ΔG, kJ/mole |
ΔG/ΔGm, % |
Длина, нт |
|
FAS |
miR-1273a |
3500 |
5'UTR |
-113 |
85 |
25 |
|
FAS |
miR-1273g-3p |
3520 |
5'UTR |
-106 |
91 |
21 |
|
FAS |
miR-466 |
2445÷2455 (5) |
5'UTR |
-102÷108 |
87÷93 |
23 |
|
FAS |
miR-4730 |
291 |
5'UTR |
-113 |
87 |
23 |
|
FDPS |
miR-4298 |
1138 |
CDS |
-110 |
88 |
22 |
|
FDPS |
miR-630 |
227 |
CDS |
-102 |
89 |
22 |
|
HSL |
miR-2114-5p |
3695 |
3'UTR |
-106 |
89 |
22 |
|
HSL |
miR-4783-5p |
2249 |
CDS |
-115 |
90 |
21 |
|
HSL |
miR-6736-5p |
506 |
CDS |
-104 |
89 |
21 |
|
HSL |
miR-6756-3p |
3516 |
3'UTR |
-104 |
91 |
20 |
|
HSL |
miR-6869-3p |
2778 |
CDS |
-113 |
90 |
21 |
|
INSIG1 |
miR-1538 |
100 |
5'UTR |
-117 |
87 |
23 |
|
LDLR |
miR-1182 |
2784 |
3'UTR |
-110 |
87 |
23 |
|
LDLR |
miR-5585-3p |
4043 |
3'UTR |
-113 |
96 |
22 |
|
LDLR |
miR-1285-5p |
4149 |
3'UTR |
-106 |
94 |
21 |
|
LDLR |
miR-1285-5p |
4322 |
3'UTR |
-102 |
91 |
21 |
|
LDLR |
miR-1285-5p |
4451 |
3'UTR |
-106 |
94 |
21 |
|
LDLR |
miR-1303 |
4159 |
3'UTR |
-106 |
91 |
22 |
|
LDLR |
miR-5095 |
3897 |
3'UTR |
-110 |
95 |
21 |
|
LDLR |
miR-619-5p |
3903 |
3'UTR |
-119 |
98 |
22 |
|
LDLR |
miR-5095 |
4372 |
3'UTR |
-104 |
89 |
21 |
|
LDLR |
miR-619-5p |
4378÷4379 (2) |
3'UTR |
-113 |
93 |
22 |
|
LDLR |
miR-619-5p |
4517 |
3'UTR |
-113 |
93 |
22 |
|
LDLR |
miR-6508-3p |
441 |
CDS |
-102 |
89 |
22 |
|
LDLR |
miR-6751-5p |
1438 |
CDS |
-115 |
90 |
23 |
|
SCAP |
miR-4656 |
1660 |
CDS |
-115 |
87 |
23 |
|
SCAP |
miR-1915-5p |
2517 |
CDS |
-113 |
88 |
22 |
|
SCAP |
miR-6763-3p |
2521 |
CDS |
-113 |
90 |
21 |
|
SCAP |
miR-3622a-5p |
3164 |
CDS |
-110 |
88 |
22 |
|
SCAP |
miR-6858-3p |
3231 |
CDS |
-113 |
88 |
22 |
|
SCAP |
miR-1178-3p |
3998 |
CDS |
-100 |
90 |
21 |
|
SREBP |
miR-4436a |
1764 |
CDS |
-102 |
89 |
21 |
|
SREBP |
miR-449b-3p |
1626 |
CDS |
-108 |
89 |
22 |
|
SREBP |
miR-4507 |
2737 |
CDS |
-106 |
91 |
20 |
|
SREBP |
miR-5197-5p |
649 |
CDS |
-102 |
87 |
23 |
|
SREBP |
miR-623 |
690 |
CDS |
-115 |
90 |
23 |
|
SREBP |
miR-6753-3p |
3079 |
CDS |
-108 |
88 |
22 |
|
SREBP |
miR-762 |
3253 |
CDS |
-121 |
89 |
22 |
|
SREBP |
miR-6756-5p |
4801 |
3'UTR |
-119 |
89 |
23 |
Однако, если концентрация miR-1273g-3p будет в несколько раз превышать концентрацию miR-1273a, то miR-1273g-3p будет большую часть времени по сравнению с miR-1273a находиться связанной с mRNA гена FAS. miR-466 имеет пять сайтов связывания в mRNA гена FAS. Эти сайты связывания расположены через два нуклеотида с наложением нуклеотидных последовательностей этих сайтов. Такие сайты связывания одной miRNA в mRNA мы назвали множественными сайтами связывания. Наличие нескольких сайтов связывания одной miRNA увеличивает вероятность связывания этой miRNA с участком mRNA содержащим множественные сайты связывания. Из пяти сайтов связывания miR-466 один сайт связывания имеет наибольшую свободную энергию взаимодействия с miR-466. Следовательно, в этой позиции miR-466 будет находиться большую часть времени, блокируя трансляцию mRNA.
Ген FDPS был мишенью только двух miRNA, сайты связывания которых располагались в CDS. mRNA гена HSL имела сайты связывания miRNA, которые располагались в CDS и 3'UTR. Эти сайты связывания не перекрывались, и каждая miRNA могла независимо связываться с mRNA гена HSL.Ген INSIG1 был мишенью всего лишь одной miRNA. Однако свободная энергия взаимодействия miR-1538 с mRNA гена INSIG1 была сравнительно высокой (-117 kJ/mole), что увеличивало способность ее связываться в 5'UTR mRNA. mRNA гена LDLR имела 12 сайтов связывания в 3'UTR и два сайта в CDS. Особенностью этого гена являлась зависимость от miR-5095 и miR-619-5p. Эти miRNA имели сайты связывания в двух участках mRNA гена LDLR, расположенных в 3'UTR на расстоянии 475 нуклеотидов. В обоих участках сайты связывания miR-5095 и miR-619-5p располагались через шесть нуклеотидов, то есть с наложением нуклеотидных последовательностей. Отметим, что miR-619-5p имела в mRNA гена LDLR четыре сайта связывания, а miR-1285-5p три сайта связывания. Величина ΔG/ΔGm для miR-1285-5p и miR-619-5p изменялась в интервале от 91% до 98%, что свидетельствует о высокой гомологии нуклеотидных последовательностей miRNA и их сайтов связывания в mRNA гена LDLR.
Все шесть miRNA связывающиеся с mRNA гена SCAP имели сайты связывания в CDS. miR-1915-5p и miR-6763-3p имели сайты связывания с наложением нуклеотидных последовательностей (таблица 1). Равные величины свободной энергии взаимодействия этих miRNA с mRNA гена SCAP предполагают конкуренцию между ними только за счет концентрации.
mRNA гена SREBP имела сайты связывания для семи miRNA в CDS и одной miRNA в 3'UTR. Все сайты связывания miRNA располагались по все длине mRNA гена SREBP без наложения нуклеотидных последовательностей.
Программа MiRTarget предсказывает взаимодействие нуклеотидов по всей длине miRNA и сайтов связывания mRNA. Для некоторых ассоциаций miRNA и mRNA генов-мишеней на рисунке 1 приведены схемы взаимодействия miRNA c mRNA генов участвующих в метаболизме липидов.
На схемах видно, что наряду с каноническими парами нуклеотидов A-U и G-C образуются водородные связи между нуклеотидами G-U и A-C. Образование таких пар, во-первых, усиливает связь между нитями miRNA и mRNA, и, во-вторых, не нарушает стэкинг-взаимодействие нуклеотидов в спиральной структуре нитей РНК.
Проведенные исследования показали, что некоторые гены, участвующие в метаболизме липидов могут служить мишенями для miRNA. Установлены ассоциации miRNA и соответствующих генов-мишеней, которые позволяют предсказывать влияние одной и более miRNA на экспрессию одного гена. В тоже время не выявлено miRNA, которые могут связываться с двумя и более генами.
Известно, что функционально важные полиморфизмы могут быть локализованы как в кодирующих, так и в некодирующих районах генов участвующих в метаболизме липидов [3]. Установленные нами сайты связывания miRNA в 5'UTR и 3'UTR позволяет предположить, что обнаруженные полиморфизмы находятся в сайтах связывания miRNA расположенных в 5'UTR и 3'UTR.
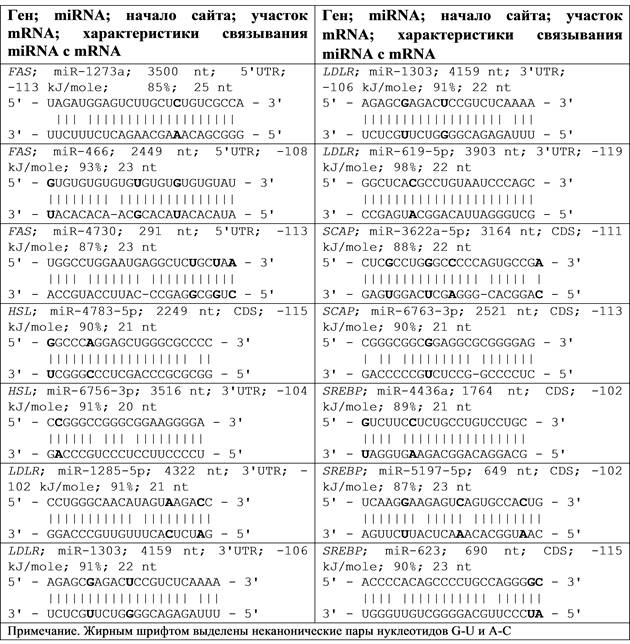
Рисунок 1. Схемы взаимодействия miRNA с mRNA генов участвующих в метаболизме липидов

