ИЗУЧЕНИЕ САЙТОВ ИНТЕГРАЦИИ КЛЕТОЧНОЙ Т-ДНК У ПРЕДСТАВИТЕЛЕЙ РАЗЛИЧНЫХ СЕКЦИЙ РОДА NICOTIANA
Секция: 2. Биологические науки
лауреатов
участников
лауреатов


участников



XV-XVI Студенческая международная заочная научно-практическая конференция «Молодежный научный форум: естественные и медицинские науки»
ИЗУЧЕНИЕ САЙТОВ ИНТЕГРАЦИИ КЛЕТОЧНОЙ Т-ДНК У ПРЕДСТАВИТЕЛЕЙ РАЗЛИЧНЫХ СЕКЦИЙ РОДА NICOTIANA
Явление горизонтального переноса от прокариот к растениям впервые было описано для бактерий рода Agrobacterium (A. rhizogenes A4) с одной стороны, и представителей рода Nicotiana (N. Glauca) — с другой [13, с. 350]. Агробактерии — это почвенные бактерии, способные переносить в клетки растений и интегрировать в растительные хромосомы фрагменты своей ДНК. Эти фрагменты называются Т-ДНК и локализуются у агробактерий на больших плазмидах Ti (у A. tumefaciens) или Ri (у A. rhizogenes). Экспрессия генов Т-ДНК приводит к развитию на растении корончатых галлов или косматых корней, которые можно рассматривать как трансгенные ткани на нетрансгенном растении. В норме привнесение Т-ДНК в растение приводит к развитию болезни и гибели растения. Однако было показано, что в геномах некоторых неинфицированных растений содержится последовательность, гомологичная Т-ДНК агробактерий. К ним относятся представители родов Nicotiana и Linaria. Т-ДНК, интегрированная в геном растений, называется клеточной Т-ДНК (клТ-ДНК). Структура последовательности: клТ-ДНК N.glauca представляет собой инвертированный повтор, содержащий открытые рамки считывания ORF11, ORF12, ORF13, и ORF14. Полученные сиквенсы повтора позволили оценить сходство между агробактериальными генами rolB и rolC, и последовательностями ORF11 и ORF12 соответственно в геноме N.glauca. Между rolB и ORF11 сходство составило 82.7 % , между rolC и ORF12 — 87.5 %. Между ORF14 и границей Т-ДНК находятся еще две открытые рамки считывания, названные NgmisL и NgmisR, гомологичные гену микимопинсинтазы mis. Было показано, что клТ-ДНК N.glauca — это интегрированная в растительный геном Т-ДНК Ri плазмиды микимопинового типа. Это было доказано сравнением полученных сиквенсов правого и левого плеча клТ-ДНК N.glauca между собой, а также с Т-ДНК четырех разных Ri плазмид: Ri1724 микимопинового типа, Ri2659 кукумопинового типа, RiА4b агропинового типа и Ri8196 маннопинового типа. Результаты сравнения показали, что сходство нуклеотидных последовательностей правого и левого плеча клТ-ДНК в районах ORF13, ORF14, и Ngmis превышают 97.9 %, а сходство аминокислотных последовательностей — 96.8 % [12, с. 778]. На данный момент неизвестно, когда произошел первый акт трансформации предков рода Nicotiana агробактериями, а также — был ли это единственный акт, или их было несколько. Для ответа на эти вопросы были исследованы различные представители рода Nicotiana. Было обнаружено, что только у двух (N. glauca, N. cordifolia) сохранился набор онкогенов, аналогичный таковому в Ri плазмиде, у N. tabacum, N. tomentosiformis, и N. otophora отсутствует ген rolB, и ещё у ряда представителей обнаружен только ген rolC (N. debneyi, N. suaveolentes, N. acuminata, N. arentsi, N. gossei, N. exigua, N. setchelli) , либо только ген rolB, изначально ошибочно принятый за rolC (N. bigelovi, N. miersi). На основе анализа последовательностей онкогенов различных штаммов агробактерий и онкогеноподобных последовательностей видов рода Nicotiana были построены филогенетические древа [7, с. 103]. Анализируя их топологию можно предположить, что Т-ДНК в геномах табаков могла появляться неоднократно. Об этом говорит то, что виды N. tabacum, N. tomentosiformis, и N. otophora имеют более высокий уровень сходства клТ-ДНК между собой и сильно отличаются от видов N. glauca, N. cordifolia, и N. debneyi (рис. 1).
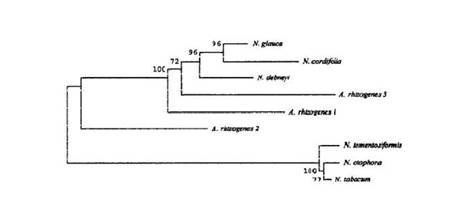
Рисунок 1. Филогенетический анализ гена rolC (Intrieri, Buiatti, 2001)
Анализ опиновых генов в составе Т-ДНК тоже может помочь прояснить детали того, каким штаммом и когда были трансформированы табаки. В работе Сузуки с соавторами методом гибридизации по Саузерну было показано, что в геномах видов N. tabacum, N. tomentosiformis, и N. otophora содержатся последовательности (rolB, rolC, ORF13, ORF14), с высокой степенью идентичности. Для N.glauca подобного показано не было. Сопоставление данных по наличию Т-ДНК в геномах и филогенетического древа, построенного традиционными методами, наводит на мысль о неоднократной трансформации представителей рода Nicotiana агробактериальной Т-ДНК. Однако в настоящее время на это счет имеются только косвенные данные. Эти данные сводятся к результатам гибридизации по Саузерну геномной ДНК различных видов Nicotiana с пограничными к Т-ДНК последовательностями N.glauca. Последние исследования клТ-ДНК у различных видов Nicotiana методом секвенирования нового поколения показали различную структуру вставок Т-ДНК, что было интерпретировано, как доказательство независимости актов трансформации [8, с. 12]. Прямым доказательством неоднократной трансформации представителей рода Nicotiana могло бы служить секвенирование пограничного с Т-ДНК района хромосомной ДНК растения.
Для выявления пограничных последовательностей ДНК в настоящее время применяют следующие методы: гибридизация по Саузерну в сочетании с ПЦР к онкогенам, ПЦР-РТ с вырожденными праймерами и зондами, а также метод «прогулка по хромосоме». В методе ПЦР-РТ с вырожденными праймерами и зондами используется набор праймеров, различающихся на пару нуклеотдов. Это позволяет проводить реакцию при неполной гомологии с матрицей. Используя метод «прогулка по хромосоме», можно анализировать протяженные участки генома при наличии сиквенса прилежащих к ним участков. Данный метод заключается в последовательном отборе клонов, содержащих частично перекрывающиеся фрагменты ДНК. Затем, с помощью маркерной ДНК проводят скриниг библиотеки клонов и отбирают подходящие. Отобранные клоны в дальнейшем служат зондами для последующего отбора, таким образом, собирается набор перекрывающихся фрагментов ДНК. Используя физическое картирование, производится упорядочивание клонов, и выстраивается последовательность [9, с. 1543].
Многими исследованиями (Ichikawa, Intrieri and Buiatti, Suzuki) было показано, что часть генов вставки, полученной в результате трансформации, экспрессируется — это может быть доказательством эволюционного значения агробактериальных генов для растительных организмов [12, с. 780]. Вставка высококонсервативна, что позволяет предположить участие полученных от бактерий генов в метаболизме растений. Было показано, что гены агробактерий влияют на биосинтез ауксинов и цитокининов в растительных клетках, меняя их соотношение, что оказывает влияние на протекание процессов регенерации и опухолеобразования [6, с. 179].
Долгое время считалось, что данный пример переноса уникален для рода Nicotiana, однако впоследствии было обнаружено наличие Т-ДНК-подобной последовательности также у представителей рода Linaria [9, с. 1549]. Присутствие такого явления у представителей различных семейств, а также наличие нескольких независимых актов трансформации разных видов одного рода может свидетельствовать о неслучайности горизонтального переноса, в связи с чем возрастает интерес к изучению данного вопроса. Целью данной работы является исследование сайтов интеграции клТ-ДНК в геном N. glauca и N. tabacum, что позволит судить о возможности неоднократных актов трансформации Nicotiana.
Материалы и методы.
Тотальная ДНК была выделена из свежих листьев асептических растений N.tabacum и N.glauca с использованием экстрагирующего буфера. Состав буфера: 0.2 г цетил-метиламмоний бромида, 0.5 мл 2М Tris-HCl (pH 8.0), 2.8 мл 5М NaCl, 0.4 мл 0.5М EDTA (pH 8.0), дистиллированная вода. Итоговый объем — 10 мл.
1.5 г свежих листьев растирали в фарфоровой ступке, затем туда был добавлен экстрагирующий буфер (10 мл), после чего содержимое ступки переносили в пробирку. Пробирку помещали в термостат на качалку в режиме 560С на 1 час. По прошествии часа в пробирку в тяге приливали 10—12 мл хлороформа, и выдерживали пробирку еще 1 час при комнатной температуре (250С), продолжая помешивать содержимое. Затем смесь центрифугировали 5 минут при 3000 об/мин и отбирали верхнюю фазу в новую пробирку. К отобранному супернатанту приливали 2/3 объема изопропанола и осторожно перемешивали, после чего оставляли пробирку при 250С на 6—8 часов. Если по прошествии этого времени был виден выпавший осадок, пробирку центрифугировали в течение 10 минут при 10.000 об/мин, сливали надосадочную жидкость и промывали осадок 70 % этанолом, после чего еще раз центрифугировали в течение 10 минут при 10.000 об/мин и сливали этанол. Осадок сушили, поместив пробирку в ламинар, затем растворяли его в дистиллированной воде.
Для амплификации интересующих генов использовалась ПЦР. Состав смеси для ПЦР: выделенная ДНК N.glauca, смесь из двух праймеров [10 пМоль/мкл] (R3R — к правой границе клТ-ДНК, L1R — к левой границе клТ-ДНК, или mis4 — к гену mis), смесь нуклеотидов [5 мМоль/мкл каждого из четырех оснований], Taq-полимераза [5 ед./мкл], десятикратный буфер на основе (NH4)2SO4, дистиллированная вода. Итоговый объем смеси составлял 50 мкл. Также проводили ПЦР на матрице выделенной ДНК N.tabacum с сочетанием праймеров LeftХR и RightХR в 9-ти возможных комбинациях. К готовой смеси добавляли 2—3 капли вазелинового масла. Все праймеры, ферменты и буфер были синтезированы компанией «Бигль».
ПЦР проводили на амплификаторе «Терцик» по следующей программе: 5 мин на 930С, 40 циклов по 15с при 930С , 30с при 500С и 30с при 720С.
Продукты амплификации разделяли в 1 % агарозном геле на однократном СБ буфере (0.4 г NaOH, 2.25 г борной кислоты на 1 л дистиллированной воды), подкрашенным бромистым этидием. Объем каждой из наносимых проб — 3 мкл. Длительность электрофореза составила 30—35 мин при напряжении 90 В. В качестве маркера молекулярного веса использовали лестницу 100 бп — 1.00 кб (1.5 мкл). Изображение геля было получено с помощью системы гель-документирования “Bio-Rad”.
Cеквенирование было выполнено на базе ресурсного центра РМИКТ. Выравнивание полученной последовательности относительно ДНК N.glauca и N.tabacum осуществляли с помощью алгоритма Clustal W и базы данных BLAST.
Последовательности использованных в работе праймеров:
1. Left1R CTGATAAGAGAAAGGATCGT
2. Left2R GGATCGTTTACTGAAAAGTCT
3. Left3R GTTTACTGAAAAGTCTTACTAC
4. Right1R TCCATTAAGGTCCTTGCGA
5. Right2R TCCTTGCGATGCCGACTGTAG
6. Right3R TGCCGACTGTAGTGCAAAATG
7. mis4 TGCTCTCGTTTGTATCGCCGTATG
Результаты и обсуждения.
В ходе данного исследования была разработана тест-система, позволяющая определить место интеграции Т-ДНК в геном представителей вида Nicotiana. Данная система представлена определенным сочетанием праймеров, захватывающих последовательность клТ-ДНК и приграничную область растительного генома. Дизайн праймеров производился с помощью последовательности N.glauca, полученной Suzuki и др. в 2002 году (Рис. 1), праймеры одной группы (Left/Right) отличаются друг от друга сдвигом захватываемой последовательности на 5—7 нуклеотидов.

Рисунок 2. Приграничная с клТ-ДНК область табачной ДНК. Выделенные области показывают идентичные участки последовательностей (Suzuki K. et al, 2002)
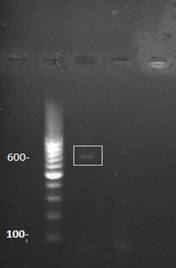
Было подобрано 3 пары праймеров и поставлена серия ПЦР, с компанованием каждого из данных шести праймеров с праймером mis4 на матрице полученной ДНК (Рис. 3). 2 из 6 праймеров показали положительный результат — в процессе реакций с ними на матрице N. glauca нарабатывался ампликон размером около 600 п. н.
На матрице ДНК N. tabacum ставили серию ПЦР с сочетанием праймеров LeftХR и RightХR в 9-ти возможных комбинациях. При использовании праймеров Left1R и Right1R в ходе реакции нарабатывалось 2 фрагмента — 350 и 400 п. н.
Размер фрагментов определили с помощью электрофореза, поставленного с полученными ПЦР-продуктами (Рис. 4).

Рисунок 3. Схема эксперимента
Т-ДНК
 |
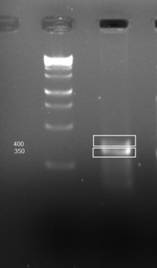 |
Рисунок 4.Фрагменты, полученные при амплификации на ДНК а) N. glauca последовательности, ограниченной праймерами mis4 и Right3R, б) N. tabacum последовательности, ограниченной праймерами Left1R и Right1R
В ходе эксперимента был получен сиквенс N. glauca, сравнение которого с последовательностью N. glauca (accession number AB071334), содержащейся в базе данных BLAST (http://blast.ncbi.nlm.nih.gov/Blast.cgi) показало, что данные последовательности совпадают, а это значит, что разработанная тест-система пригодна для использования. Используя полученную систему, удалось показать, что место интеграции Т-ДНК у N. glauca отличается от такового у N. tabacum, поскольку не удалось наработать фрагмента на ДНК N. tabacum ни с одной комбинацией mis4-LeftXR, mis4-RightXR. В то же время отмечено образование ПЦР продукта при использовании комбинации праймеров LeftXR- RightXR. Короткий размер ампликона свидетельствует о том, что между сайтами посадки данных праймеров нет клТ-ДНК, а сиквенс фрагмента показывает его сходство с пограничной к кл-Т-ДНК последовательностью N.glauca (Рис. 5).

Рисунок 5. Выравнивание нуклеотидных последовательностей фрагмента полученного при амплификации на матрице N. tabacum последовательности, ограниченной праймерами Left1R и Right1R, и приграничной к клТ-ДНК последовательности N. glauca, (accession number AB071334)
Заключение.
В ходе данной работы была разработана тест-система, показавшая себя как удобный и надежный метод для поиска сайта интеграции Т-ДНК в растительный геном, что было доказано полученным сиквенсом N.glauca. Применение данного метода в исследованиях N. tabacum показало, что место интеграции Т-ДНК в геном N. tabacum отличается от такового у N.glauca. Планируется проведение дальнейших исследований, включающих поиск сайта интеграции у N. tabacum, что позволило бы скринировать другие виды табака в отношении сайтов интеграции клТ-ДНК и сделать вывод о количестве актов трансформации агробактериями представителей данного рода в ходе его эволюции.
Данная работа была выполнена при поддержке гранта РФФИ №14-04-01480 А, темпланов СПбГУ 1.39.315.2014, 0.37.526.2013.
Список литературы:
1. Алгоритм для выравнивания последовательностей — [Электронный ресурс] — URL: http://align.genome.jp.
2. База данных National Center for Biotechnology Information — [Электронный ресурс] — URL: http://blast.ncbi.nlm.nih.gov/Blast.cgi.
3. О.А. Кулаева, Т.В. Матвеева, Л.А. Лутова Горизонтальный перенос генов от агробактерий к растениям. Экологическая генетика 2006. Т. IV. № 4. С. 10—19.
4. Michael Webster Bevan and Mary-Dell Chilton T-DNA of the Agrobacterium Ti and Ri plasmids. Ann. Rev. Genet 1982. 16:3S7—4.
5. Furner I.J., Huffman G.A., Amasino R.M. [et al.] An Agrobacterium transformation in the evolution of the genus Nicotiana. Nature 1986. Vol. 319. P. 422—427.
6. Ichikawa T., Ozeki Y., Syono K. Evidence for the expression of the rol genes of N. glauca in genetic tumors of N. glauca x N. langsdorffii. Mol. Gen. Genet. 1990. Vol. 220, № 2. P. 177—180.
7. Intrieri M.C., Buiatti M. The horizontal transfer of Agrobacterium rhizogenes genes and the evolution of the genus Nicotiana. Molecular Phylogenetics and evolution. 2001. Vol. 20. P. 100—110.
8. Ke Chen, François Dorlhac de Borne, Ernö Szegedi, Léon Otten. Deep sequencing of the ancestral tobacco species Nicotiana tomentosiformis reveals multiple T-DNA inserts and a complex evolutionary history of natural transformation in the genus Nicotiana. Plant J. 2014. Sep 15. doi: 10.1111/tpj.12661.
9. Matveeva T.V., Bogomaz D.I., Pavlova O.A., Nester E.W., Lutova L.A. Horizontal Gene Transfer from Genus Agrobacterium to the Plant Linaria in Nature . 2012. MPMI Vol. 25, № 12, P. 1542—1551.
10. Murashige T., F. Skoog. A revised medium for rapid growth and bioassays with tobacco cultures. 1962. Physiol. Plant 15:473—497.
11. Sambrook J., Russell D.W. Molecular Cloning: a laboratory manual. N.Y. 2001.
12. Suzuki K, Yamashita I., Tanaka N. Tobacco plants were transformed by Agrobacterium rhizogenes infection during their evolution. 2002. Plant J 32(5):775—787.
13. White F.F., Garfinkel D.J., Huffman G.A., Gordon M.P., and Nester E.W. Sequence homologous to Agrobacterium rhizogenes T- DNA in the genomes of uninfected plants. 1983. Nature 301:348—350.

