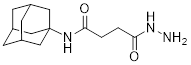
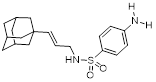РАЦИОНАЛЬНЫЙ ДИЗАЙН ИНГИБИТОРОВ ГЕЛИКАЗЫ NSP13 КОРОНАВИРУСОВ
Конференция: XLIX Международная научно-практическая конференция «Научный форум: медицина, биология и химия»
Секция: Медицинская химия

XLIX Международная научно-практическая конференция «Научный форум: медицина, биология и химия»
РАЦИОНАЛЬНЫЙ ДИЗАЙН ИНГИБИТОРОВ ГЕЛИКАЗЫ NSP13 КОРОНАВИРУСОВ
RATIONAL DESIGN OF CORONAVIRAL HELICASE NSP13 INHIBITORS
Vadim Shiryaev
Candidate of Chemical Sciences, docent at Department of organic chemistry Samara State Technical University, Russia, Samara
Аннотация. Проведен обзор потенциальных мишеней коронавирусов и для одной из потенциальных мишеней – геликазы Nsp13 – проведен молекулярный докинг для выявления ее потенциальных ингибиторов.
Abstract. A review of potential targets of coronaviruses was carried out, and for one of the potential targets, Helicase Nsp13, molecular docking was carried out to identify its potential inhibitors.
Ключевые слова: коронавирус; геликаза; Nsp13; каркасные соединения.
Keywords: coronavirus; Helicase, Nsp13; cage compounds.
В настоящее время особенно серьезной проблемой в медицине является терапия вирусных заболеваний. К 2020 г. всего зарегистрировано 93 противовирусных препарата, направленных на лечение заболеваний, вызываемых только лишь 9 вирусами: вирусом иммунодефицита человека (ВИЧ), вирусом гепатита В, вирусом гепатита С, вирусом герпеса, вирусом гриппа, цитомегаловирусом, вирусом варицелла-зостер, респираторно-синцитиальным вирусом и вирусом папилломы человека [1, 2].
До настоящего времени поиски препаратов направленного действия против коронавирусов не принесли положительных результатов. Несмотря на возникновение новых штаммов коронавирусов, приводящих к более легкому течению заболевания, и наличие действующих вакцин, вероятность возникновения новых вспышек тяжелого острого респираторного синдрома (ТОРС / SARS) вследствие вероятности снижения эффективности вакцин из-за мутации вируса, а также терапия пациентов, вакцинация которых по невозможна, требуют поиска химиотерапевтических препаратов, способных воздействовать на коронавирусы.
В настоящее время не существует химиотерапевтических препаратов, обладающих подтвержденной активностью in vivo в отношении коронавирусов, в том числе штамма, вызвавшего текущую пандемию SARS2-CoV-2 (2019-nCoV). Среди исследователей преобладают два основных направления поиска: перепрофилирование (перепозиционирование) существующих лекарственных препаратов и дизайн низкомолекулярных ингибиторов функций вирусных и вовлеченных клеточных белков. На настоящий момент для 20 потенциальных мишеней известна трехмерная структура. Наиболее перспективными точками вмешательства в репродуктивный цикл в настоящее время считаются: химотрипсин-подобная протеиназа 3cpro/Mpro/nsp5 [4], папаин-подобная протеиназа PLpro [5], ионный канал E [6], поверхностный белок S/spike [7], вирусная геликаза nsp13 [8], митоген-активированная протеин киназа и РНК-зависимая РНК-полимераза RdRp [9].
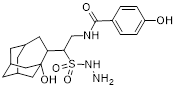
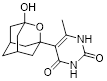
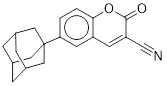
Среди других перспективных вирусных мишеней имеется класс белков, принимающих участие в репликации ДНК и трансляции РНК – геликазы [Nat. Rev. Drug Disc., 2005, 4, 845–853]. Основной функцией геликаз является разделение цепей двухцепочечной молекулы ДНК или внутримолекулярных связей в молекулах РНК, используя энергию гидролиза АТФ или ГТФ. Коронавирусы также имеют ген, кодирующий геликазу nsp13 [8, 11], для которой известна трехмерная модель комплекса транскрипции [12], а также имеются модели комплексов геликазы с серией малых молекул (записи 5RL6-5RL9, 5RLB-5RLW, 5RLY-5RLZ, 5RM0-5RM9, 5RMA-5RMM из базы данных RCSB PDB). Для данной геликазы in vitro обнаружен ряд ингибиторов – производных оксазолопиридина [13], порфиринового комплекса висмута [14], 1,2,4-триазола [15], бананина [16, 17], производных дигидрохромона [18-20] и арилдикетокислот [21]
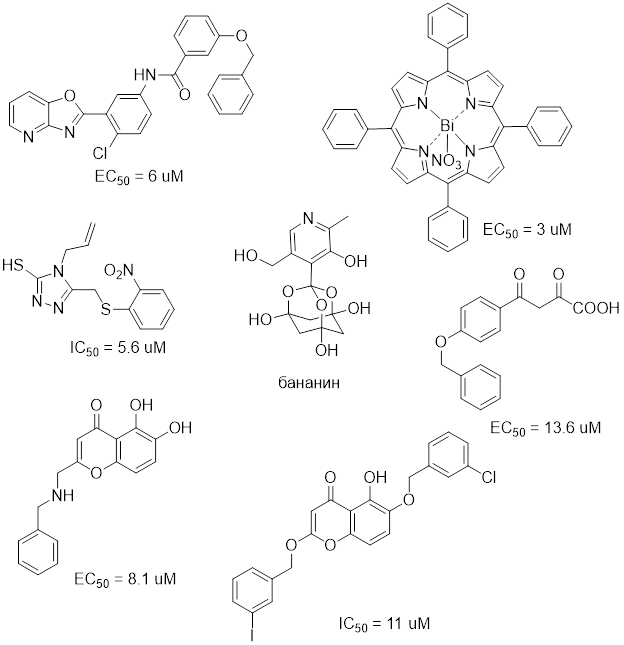
Рисунок 1. Некоторые ингибиторы геликазы Nsp13 коронавирусов
Под формулами указаны полуингибирующие концентрации в мкМ/л.
В соответствии с экспериментальными данными мы предприняли попытку виртуального дизайна потенциальных ингибиторов коронавирусной геликазы Nsp13. В качестве модели была выбраны трехмерная структура 6JYT из банка данных трёхмерных структур белков и нуклеиновых кислот PDB, определенная при помощи рентгеноструктурного анализа.
В качестве возможных лигандов была генерирована серия соединений, содержащих фрагменты адамантан, гомоадамантана и бицикло[3.3.1]нонана и имеющих остатка карбо и гетероциклов различной степени замещенности. Общий размер виртуальной библиотеки составил 250 соединений. В качестве сайта связывания бы выбран карман, предназначенные для размещения одной из цепей РНК. Результаты докинга базы виртуальных соединений позволили выбрать несколько структур, обладающих наибольшими расчетными энергиями связывания с геликазой. Анализ взаимодействий лиганд белок показал, что чаще всего возникают контакты с аминокислотными остатками пролина 284, глутамина 375, метионина 378, глутамина 404, и аргинина 567 (Таблица 1). Область связывания лигандов и важнейшие аминокислотные остатки показаны на рисунке 2.
Таблица 1.
Результаты молекулярного докинга виртуальной базы соединений в сайт связывания геликазы nsp13 и наиболее важные для связывания аминокислотные остатки
|
№ |
Структура |
Оценочная энергия связывания, кДж/моль |
Взаимодействия с белком |
|---|---|---|---|
|
1 |
|
-8,3 |
Lys288, Glu375, Met378, Gln404, Arg567 |
|
2 |
|
-7,9 |
Gly282, Pro284, Cys288, Glu375, Gly538, Arg567 |
|
3 |
|
-7,6 |
Gly538, Glu540, Arg567 |
|
4 |
|
-7,6 |
Pro284, Glu375, Gln404, Gln537 |
|
5 |
|
-7,6 |
Gly285, Met378, Gln404, Gln537, Glu540, Arg567 |
|
6 |
|
-7,2 |
Pro284, Gly285, Lys288, Glu375, Asp401 |
|
7 |
|
-7,1 |
Met378, Gln404, Arg567 |
|
8 |
|
-7,1 |
Pro284, Arg567 |
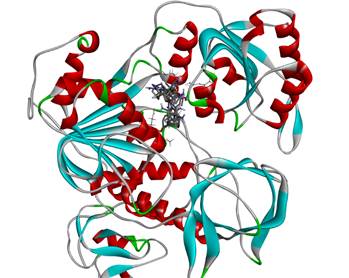
Рисунок 2. Наложение лигандов, показавших лучшие энергии связывания, в сайте геликазы Nsp13
Экспериментальная часть. Подготовка лигандов. Структуры лигандов проходили первоначальную оптимизацию при помощи программного пакета Avogadro 1.2.1 в силовом поле MMFF94. Полученные трехмерные модели конвертировались в формат, подходящий для программы докинга AutoDock Vina при помощи пакета визуализации AutoDock Tools.
Подготовка белка. Трехмерная модель 6JYT была получена из банка данных трёхмерных структур белков и нуклеиновых кислот PDB и проверена на наличие ошибок в расположении тяжелых атомов и позиций водорода. Из модели была удалена вода и низкомолекулярные соединения, и получившаяся структура конвертировалась в формат, подходящий для программы докинга AutoDock Vina при помощи пакета визуализации AutoDock Tools.
Проведение молекулярного докинга и анализ результатов. В качестве места для проведения молекулярного докинга был выбран бокс размером 20х25х20 Å с координатами центра -16.98 Å, -0.21 Å и 0.84 Å. Отбор наилучших соединений производился по значениям расчетной энергии связывания меньше 7,1 кДж/моль. Визуализация контактов лиганд-аминокислота и общего расположения лиганда в кармане связывания производилось при помощи пакета Biovia Discovery Studio 2019.
Вывод. На основе виртуальной модели геликазы Nsp13 коронавирусов при помощи молекулярного докинга были определены потенциальные ингибиторы работы данного белка и определены критические виды взаимодействия белок-лиганд для проявления эффективного связывания потенциального лекарственного препарата с мишенью.
Работа выполнена при финансовой поддержке Российского фонда фундаментальных исследований (проект РФФИ 20-53-04035).