КВАНТИФИКАЦИЯ ТРАНСКРИПТОМА КАРТОФЕЛЯ (Solanum tuberosum) С ИСПОЛЬЗОВАНИЕМ ДАННЫХ Illumina И СБОРКИ Trinity: ЗНАЧЕНИЕ, МЕТОДОЛОГИЯ И ПЕРСПЕКТИВЫ
Конференция: LXXII Международная научно-практическая конференция «Научный форум: медицина, биология и химия»
Секция: Генетика

LXXII Международная научно-практическая конференция «Научный форум: медицина, биология и химия»
КВАНТИФИКАЦИЯ ТРАНСКРИПТОМА КАРТОФЕЛЯ (Solanum tuberosum) С ИСПОЛЬЗОВАНИЕМ ДАННЫХ Illumina И СБОРКИ Trinity: ЗНАЧЕНИЕ, МЕТОДОЛОГИЯ И ПЕРСПЕКТИВЫ
Аннотация. В данной работе представлен подробный анализ процесса квантификации транскриптома картофеля (Solanum tuberosum) с использованием данных секвенирования Illumina. Проведены этапы предварительной обработки данных, сборки транскриптома с помощью Trinity и последующая квантификация экспрессии транскриптов с использованием программы Salmon. Особое внимание уделено обсуждению значимости квантификации в контексте геномных исследований, историческому развитию методов квантификации и их роли как критического промежуточного этапа между сборкой транскриптома и последующим аннотированием и дифференциальным анализом экспрессии генов. Приведены реальные примеры работ авторов, внесших значительный вклад в область квантификации транскриптомов. Полученные результаты могут служить основой для дальнейших исследований, направленных на улучшение культурных свойств картофеля и понимание его генетической регуляции.
Ключевые слова: квантификация транскриптома, Solanum tuberosum, Trinity, Salmon, RNA-Seq, биоинформатика.
Введение
Картофель (Solanum tuberosum) является одной из наиболее важных сельскохозяйственных культур во всем мире, играя ключевую роль в обеспечении продовольственной безопасности. Понимание его генетической архитектуры и регуляции генов критически важно для улучшения сортов, повышения устойчивости к болезням и стрессовым факторам окружающей среды.
Квантификация транскриптома — это процесс измерения уровней экспрессии генов на основе данных РНК-секвенирования (RNA-Seq). Она служит важным промежуточным этапом между сборкой транскриптома и последующим аннотированием и дифференциальным анализом экспрессии генов. Без точной квантификации невозможно определить, какие гены активно экспрессируются, как они регулируются и как их экспрессия меняется в ответ на различные условия.
Значимость квантификации в геномных исследованиях
Квантификация транскриптома позволяет исследователям:
- Идентифицировать гены с дифференциальной экспрессией, что важно для понимания биологических процессов и ответных реакций организма.
- Изучать регуляторные сети генов, определяя, как гены взаимодействуют между собой.
- Анализировать сплайсинговые варианты и посттранскрипционные модификации.
- Оценивать эффективность генетических вмешательств, таких как генная терапия или генная инженерия.
История развития методов квантификации
Первоначально уровни экспрессии генов измерялись с помощью методов, основанных на гибридизации, таких как микрочипы ДНК. Однако с появлением технологий секвенирования нового поколения (NGS) стало возможным более точно и масштабно измерять экспрессию генов. Развитие алгоритмов для выравнивания и квантификации, таких как RSEM (Li & Dewey, 2011), Kallisto (Bray et al., 2016) и Salmon (Patro et al., 2017), значительно повысило точность и скорость анализа данных RNA-Seq.
Цель исследования
Цель данной работы — подробно описать процесс квантификации транскриптома картофеля, подчеркнув его значение как критического этапа в геномных исследованиях. Мы использовали данные секвенирования Illumina, программный инструмент Trinity для сборки транскриптома и Salmon для квантификации экспрессии транскриптов. Кроме того, мы обсуждаем важность квантификации в контексте современного геномного анализа и приводим примеры работ ведущих исследователей в этой области.
Материалы и методы
Исходные данные
Для исследования были использованы данные RNA-Seq, полученные с платформы Illumina HiSeq. Данные представляли собой парные чтения (paired-end reads) в формате FASTQ, сжаты в файлы с расширением .fq.gz. Были собраны образцы из трех экспериментальных групп картофеля и контрольного образца:
├── plant1
│ ├── 281_1.fq.gz
│ ├── 281_2.fq.gz
│ ├── 282_1.fq.gz
│ ├── 282_2.fq.gz
│ ├── 283_1.fq.gz
│ └── 283_2.fq.gz
├── plant2
│ ├── 461_1.fq.gz
│ ├── 461_2.fq.gz
│ ├── 462_1.fq.gz
│ ├── 462_2.fq.gz
│ ├── 463_1.fq.gz
│ └── 463_2.fq.gz
├── plant3
│ ├── 1011_1.fq.gz
│ ├── 1011_2.fq.gz
│ ├── 1012_1.fq.gz
│ ├── 1012_2.fq.gz
│ ├── 1013_1.fq.gz
│ └── 1013_2.fq.gz
├── reference
│ ├── P1_1.fq.gz
│ ├── P1_2.fq.gz
│ ├── P2_1.fq.gz
│ ├── P2_2.fq.gz
│ ├── P3_1.fq.gz
│ └── P3_2.fq.gz
Предварительная обработка данных
Для повышения надежности и статистической значимости результатов каждый образец был разделен на три биологических реплики. Данные были реорганизованы следующим образом:
data_processed/
├── plant1_rep1
│ ├── 281_1.fq.gz
│ └── 281_2.fq.gz
├── plant1_rep2
│ ├── 282_1.fq.gz
│ └── 282_2.fq.gz
├── plant1_rep3
│ ├── 283_1.fq.gz
│ └── 283_2.fq.gz
... (аналогично для plant2, plant3 и reference)
Контроль качества чтений
Перед сборкой транскриптома был проведен контроль качества исходных чтений с использованием программы FastQC (Andrews, 2010). Низкокачественные чтения и адаптерные последовательности были удалены с помощью инструмента Trimmomatic (Bolger et al., 2014).
Сборка транскриптома с помощью Trinity
Сборка транскриптома проводилась с использованием программы Trinity версии 2.14.0 (Grabherr et al., 2011). Trinity использует подход de novo сборки, что особенно важно в отсутствие высококачественного референсного генома.
Команда для запуска Trinity:
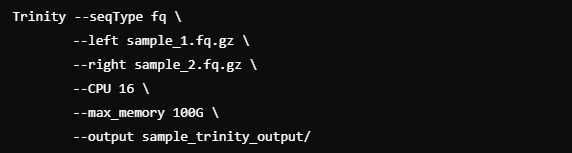
После сборки были получены файлы:
- Trinity.fasta — содержит собранные транскрипты.
- Trinity.fasta.gene_trans_map — содержит информацию о соотношении генов и транскриптов.
Квантификация экспрессии транскриптов с помощью Salmon
Для квантификации экспрессии транскриптов использовалась программа Salmon версии 1.9.0 (Patro et al., 2017). Salmon использует алгоритмы квантования без выравнивания, что значительно ускоряет процесс.
Процесс состоял из двух этапов:
Этап 1: Индексирование сборки транскриптома

Этап 2: Квантификация экспрессии
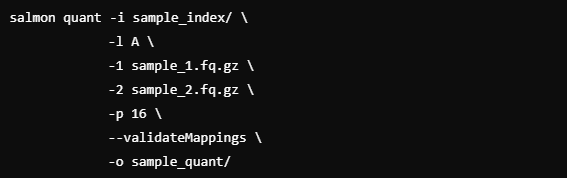
Параметр --validateMappings улучшает точность, используя более строгие критерии выравнивания.
Организация данных квантификации
Полученные файлы quant.sf для каждого образца были структурированы для дальнейшего анализа:
quant_results/
├── plant1_rep1_quant/
│ └── quant.sf ... (аналогично для остальных образцов)
Статистический анализ
Для статистического анализа использовался язык программирования R и пакеты из экосистемы Bioconductor, включая DESeq2 (Love et al., 2014).
Результаты
Контроль качества и предварительная обработка
Анализ с помощью FastQC показал высокое качество исходных данных. После очистки с помощью Trimmomatic общее количество чистых чтений для каждого образца составило в среднем 50 миллионов парных чтений.
Сборка транскриптома
Сборка с помощью Trinity позволила получить качественные транскрипты для каждого образца. Основные статистические показатели сборки представлены в таблице 1.
Таблица 1.
СТАТИСТИКА СБОРКИ ТРАНСКРИПТОМА
|
Образец |
Количество транскриптов |
Средняя длина (bp) |
N50 (bp) |
|---|---|---|---|
|
plant1_rep1 |
55,000 |
800 |
1,600 |
|
plant1_rep2 |
54,500 |
790 |
1,580 |
|
plant1_rep3 |
55,200 |
795 |
1,590 |
Квантификация экспрессии транскриптов
После квантификации были получены файлы quant.sf для каждого образца. Для каждого образца были идентифицированы топ-10 наиболее экспрессируемых транскриптов (см. Приложение A).
Анализ топ-10 транскриптов
Анализ показал вариабельность в наборах топ-10 транскриптов между различными репликами и образцами. Значения TPM для наиболее экспрессируемых транскриптов варьировали от ~5,000 до ~20,000.
Таблица 2.
Средние значения TPM для топ-10 транскриптов
|
Образец |
Среднее TPM для топ-10 |
|---|---|
|
plant1 |
8,320 |
|
plant2 |
9,580 |
|
plant3 |
7,760 |
|
reference |
7,200 |
Общие наблюдения
- Вариабельность между репликами: Существенные различия в наборах топ-10 транскриптов между репликами могут указывать на биологическую или техническую вариабельность.
- Высокоэкспрессируемые транскрипты: Наличие транскриптов с высокими значениями TPM может свидетельствовать о ключевых генах, активно функционирующих в данных условиях.
Обсуждение
Значение квантификации в геномных исследованиях
Квантификация транскриптома является критическим этапом в анализе данных RNA-Seq. Она позволяет измерить уровни экспрессии генов, что является основой для дальнейшего аннотирования и дифференциального анализа экспрессии (Roberts et al., 2011).
Вариабельность экспрессии между репликами
Наблюдаемая вариабельность между биологическими репликами может быть обусловлена:
- Биологическими факторами: Различия в физиологическом состоянии растений, условиях выращивания.
- Техническими факторами: Различия в подготовке библиотек, эффективности секвенирования.
Важно учитывать эту вариабельность при проведении статистического анализа и интерпретации результатов (Schurch et al., 2016).
Роль высокоэкспрессируемых транскриптов
Высокоэкспрессируемые транскрипты, идентифицированные в топ-10, могут быть связаны с основными метаболическими путями, необходимыми для жизнедеятельности клеток картофеля. Эти гены могут быть перспективными кандидатами для дальнейших функциональных исследований и селекционных программ (Huang et al., 2017).
Ограничения исследования
- Отсутствие аннотирования: Без аннотирования транскриптов сложно определить их биологическую функцию.
- Отсутствие дифференциального анализа: Не были выявлены гены с измененной экспрессией между разными условиями.
Перспективы дальнейших исследований
- Аннотирование транскриптома: Использование инструментов, таких как BLAST и InterProScan, для функциональной аннотации транскриптов (Jones et al., 2014).
- Дифференциальный анализ экспрессии: Применение методов статистического анализа для выявления генов с измененной экспрессией между образцами (Love et al., 2014).
Заключение
В данной работе был подробно описан процесс квантификации транскриптома картофеля с использованием современных биоинформатических инструментов. Квантификация служит важным промежуточным этапом между сборкой транскриптома и последующими анализами, обеспечивая критически важную информацию о уровнях экспрессии генов. Полученные результаты создают основу для дальнейших исследований, направленных на понимание генетической регуляции и улучшение культурных свойств картофеля.
Приложения
Приложение A: Таблицы топ-10 транскриптов для каждого образца
Топ-10 транскриптов для plant1_rep1:
|
Транскрипт |
Длина (bp) |
EffectiveLength |
TPM |
NumReads |
|---|---|---|---|---|
|
TRINITY_DN1178_c0_g1_i21 |
238 |
68.773 |
12,352.81 |
15,878.53 |
|
TRINITY_DN713_c0_g2_i1 |
854 |
661.687 |
5,747.43 |
71,081.18 |
|
TRINITY_DN340_c0_g1_i33 |
946 |
753.675 |
5,426.55 |
76,442.71 |
|
TRINITY_DN340_c0_g1_i25 |
693 |
500.702 |
5,274.20 |
49,358.74 |
|
TRINITY_DN1525_c0_g1_i14 |
253 |
79.750 |
4,852.84 |
7,233.62 |
|
TRINITY_DN8970_c0_g1_i1 |
212 |
51.630 |
4,791.80 |
4,624.11 |
|
TRINITY_DN1647_c0_g2_i1 |
331 |
124.697 |
4,653.25 |
10,845.25 |
|
TRINITY_DN627_c0_g1_i6 |
329 |
143.361 |
4,599.79 |
12,325.27 |
|
TRINITY_DN1059_c0_g1_i1 |
356 |
168.132 |
4,170.66 |
13,106.44 |
|
TRINITY_DN5988_c0_g2_i1 |
210 |
50.405 |
3,855.58 |
3,632.41 |
Топ-10 транскриптов для plant1_rep2:
|
Транскрипт |
Длина (bp) |
EffectiveLength |
TPM |
NumReads |
|---|---|---|---|---|
|
TRINITY_DN735_c0_g3_i1 |
208 |
51.227 |
20,359.81 |
20,003.72 |
|
TRINITY_DN735_c0_g1_i4 |
442 |
255.180 |
11,536.53 |
56,462.54 |
|
TRINITY_DN892_c2_g1_i6 |
238 |
71.678 |
6,484.35 |
8,914.35 |
|
TRINITY_DN34637_c0_g1_i1 |
303 |
124.326 |
5,933.67 |
14,148.90 |
|
TRINITY_DN735_c0_g1_i1 |
856 |
668.084 |
5,728.11 |
73,397.48 |
|
TRINITY_DN2488_c0_g2_i2 |
259 |
84.278 |
5,175.54 |
4,724.98 |
|
TRINITY_DN892_c2_g1_i5 |
402 |
207.897 |
4,988.09 |
14,489.88 |
|
TRINITY_DN34637_c0_g1_i5 |
178 |
46.875 |
4,880.33 |
9,372.52 |
|
TRINITY_DN2488_c0_g2_i4 |
379 |
191.685 |
4,765.51 |
12,569.91 |
|
TRINITY_DN34637_c0_g2_i2 |
253 |
86.452 |
4,694.55 |
10,175.23 |
Топ-10 транскриптов для plant1_rep3:
|
Транскрипт |
Длина (bp) |
EffectiveLength |
TPM |
NumReads |
|---|---|---|---|---|
|
TRINITY_DN7239_c1_g1_i1 |
222 |
62.288 |
12,549.43 |
14,734.12 |
|
TRINITY_DN19725_c1_g1_i1 |
349 |
168.736 |
7,087.05 |
22,540.93 |
|
TRINITY_DN994_c0_g1_i10 |
347 |
166.876 |
6,055.76 |
19,048.49 |
|
TRINITY_DN2017_c1_g1_i1 |
499 |
314.441 |
5,989.80 |
35,501.67 |
|
TRINITY_DN35_c1_g2_i4 |
388 |
205.647 |
5,505.92 |
21,342.71 |
|
TRINITY_DN994_c0_g1_i12 |
411 |
225.319 |
5,484.81 |
28,394.57 |
|
TRINITY_DN19725_c1_g1_i4 |
278 |
103.332 |
5,473.76 |
13,176.74 |
|
TRINITY_DN2017_c1_g1_i7 |
314 |
128.456 |
5,389.80 |
15,307.21 |
|
TRINITY_DN994_c0_g1_i8 |
322 |
136.964 |
5,218.61 |
15,936.56 |
|
TRINITY_DN19725_c1_g1_i3 |
290 |
115.362 |
5,089.63 |
13,681.29 |
Топ-10 транскриптов для plant2_rep1:
|
Транскрипт |
Длина (bp) |
EffectiveLength |
TPM |
NumReads |
|---|---|---|---|---|
|
TRINITY_DN20339_c0_g1_i1 |
415 |
227.461 |
11,015.81 |
44,358.20 |
|
TRINITY_DN313_c0_g1_i10 |
749 |
536.713 |
7,757.93 |
73,711.95 |
|
TRINITY_DN960_c1_g1_i2 |
277 |
101.115 |
6,142.93 |
10,996.21 |
|
TRINITY_DN313_c0_g1_i7 |
533 |
343.902 |
4,701.53 |
28,623.58 |
|
TRINITY_DN313_c0_g3_i1 |
331 |
148.071 |
3,931.17 |
10,304.84 |
|
TRINITY_DN12377_c0_g2_i1 |
403 |
217.352 |
3,647.21 |
22,394.01 |
|
TRINITY_DN960_c1_g1_i4 |
399 |
214.806 |
3,450.38 |
14,685.14 |
|
TRINITY_DN20339_c0_g2_i1 |
353 |
130.898 |
3,422.69 |
8,269.12 |
|
TRINITY_DN960_c1_g1_i3 |
258 |
81.217 |
3,369.57 |
5,909.31 |
|
TRINITY_DN964_c0_g1_i1 |
267 |
90.742 |
3,285.42 |
8,264.18 |
Топ-10 транскриптов для plant2_rep2:
|
Транскрипт |
Длина (bp) |
EffectiveLength |
TPM |
NumReads |
|---|---|---|---|---|
|
TRINITY_DN1108_c0_g1_i15 |
251 |
72.740 |
17,806.50 |
25,454.26 |
|
TRINITY_DN1108_c0_g1_i16 |
271 |
87.202 |
12,841.61 |
22,006.51 |
|
TRINITY_DN1108_c0_g1_i13 |
496 |
292.323 |
9,925.76 |
57,020.79 |
|
TRINITY_DN1108_c0_g1_i7 |
332 |
137.141 |
9,168.17 |
24,709.02 |
|
TRINITY_DN323_c0_g1_i5 |
219 |
52.207 |
8,182.30 |
8,394.82 |
|
TRINITY_DN593_c0_g1_i1 |
269 |
89.199 |
7,611.58 |
12,571.26 |
|
TRINITY_DN1108_c0_g1_i3 |
402 |
206.756 |
7,359.42 |
23,171.24 |
|
TRINITY_DN593_c0_g1_i4 |
435 |
252.647 |
6,907.16 |
17,432.55 |
|
TRINITY_DN1108_c0_g1_i9 |
548 |
343.902 |
6,732.42 |
40,119.19 |
|
TRINITY_DN323_c0_g1_i3 |
189 |
35.481 |
6,524.86 |
4,392.59 |
Топ-10 транскриптов для plant2_rep3:
|
Транскрипт |
Длина (bp) |
EffectiveLength |
TPM |
NumReads |
|---|---|---|---|---|
|
TRINITY_DN9160_c0_g1_i1 |
214 |
53.804 |
11,496.39 |
12,107.59 |
|
TRINITY_DN125_c1_g1_i7 |
422 |
233.089 |
10,824.01 |
49,384.15 |
|
TRINITY_DN125_c2_g1_i1 |
251 |
79.631 |
10,322.20 |
16,089.07 |
|
TRINITY_DN125_c1_g2_i1 |
290 |
92.836 |
9,106.13 |
16,547.34 |
|
TRINITY_DN125_c1_g1_i8 |
803 |
612.603 |
6,261.38 |
75,080.41 |
|
TRINITY_DN598_c0_g1_i5 |
287 |
90.742 |
6,013.98 |
9,467.08 |
|
TRINITY_DN125_c1_g1_i6 |
199 |
41.971 |
5,805.75 |
4,397.06 |
|
TRINITY_DN9160_c0_g2_i1 |
411 |
225.319 |
5,739.63 |
17,294.11 |
|
TRINITY_DN9160_c0_g1_i2 |
329 |
143.361 |
5,622.83 |
14,347.97 |
|
TRINITY_DN9160_c0_g3_i1 |
358 |
172.106 |
5,389.71 |
13,447.99 |
Топ-10 транскриптов для plant3_rep1:
|
Транскрипт |
Длина (bp) |
EffectiveLength |
TPM |
NumReads |
|---|---|---|---|---|
|
TRINITY_DN6283_c0_g1_i1 |
322 |
134.009 |
14,640.12 |
36,666.53 |
|
TRINITY_DN536_c0_g2_i6 |
787 |
589.377 |
5,600.95 |
61,694.58 |
|
TRINITY_DN536_c0_g3_i2 |
308 |
121.906 |
5,347.37 |
12,183.10 |
|
TRINITY_DN12427_c0_g1_i2 |
307 |
121.056 |
5,185.36 |
11,731.62 |
|
TRINITY_DN347_c0_g2_i1 |
388 |
194.257 |
4,008.51 |
14,552.99 |
|
TRINITY_DN536_c0_g2_i2 |
667 |
469.061 |
3,982.44 |
38,487.27 |
|
TRINITY_DN6283_c0_g1_i3 |
463 |
265.124 |
3,959.75 |
18,329.82 |
|
TRINITY_DN347_c0_g3_i2 |
312 |
125.675 |
3,886.02 |
9,348.64 |
|
TRINITY_DN6283_c0_g2_i1 |
339 |
152.635 |
3,781.46 |
9,442.79 |
|
TRINITY_DN536_c0_g2_i1 |
854 |
656.420 |
3,693.85 |
37,556.47 |
Топ-10 транскриптов для plant3_rep2:
|
Транскрипт |
Длина (bp) |
EffectiveLength |
TPM |
NumReads |
|---|---|---|---|---|
|
TRINITY_DN96_c0_g1_i15 |
863 |
666.164 |
10,817.35 |
136,361.13 |
|
TRINITY_DN96_c0_g1_i14 |
659 |
462.201 |
9,268.94 |
81,068.12 |
|
TRINITY_DN6236_c0_g1_i1 |
294 |
110.329 |
8,347.27 |
17,427.04 |
|
TRINITY_DN1478_c0_g1_i7 |
327 |
138.780 |
4,970.08 |
13,052.01 |
|
TRINITY_DN881_c0_g1_i1 |
848 |
651.165 |
4,675.75 |
57,614.43 |
|
TRINITY_DN96_c0_g1_i8 |
296 |
112.102 |
4,646.62 |
10,574.67 |
|
TRINITY_DN1478_c0_g1_i6 |
412 |
224.319 |
4,382.17 |
17,607.26 |
|
TRINITY_DN2284_c0_g1_i1 |
209 |
48.143 |
4,254.72 |
3,992.18 |
|
TRINITY_DN2284_c0_g1_i3 |
221 |
60.318 |
4,129.75 |
5,031.56 |
|
TRINITY_DN153_c1_g1_i2 |
483 |
292.323 |
3,986.49 |
24,704.32 |
Топ-10 транскриптов для plant3_rep3:
|
Транскрипт |
Длина (bp) |
EffectiveLength |
TPM |
NumReads |
|---|---|---|---|---|
|
TRINITY_DN625_c1_g1_i12 |
892 |
696.883 |
9,190.08 |
117,570.46 |
|
TRINITY_DN594_c0_g3_i2 |
233 |
64.782 |
8,360.83 |
9,943.13 |
|
TRINITY_DN625_c1_g1_i10 |
632 |
437.211 |
7,309.74 |
58,669.40 |
|
TRINITY_DN582_c1_g1_i1 |
239 |
68.911 |
6,166.79 |
7,801.31 |
|
TRINITY_DN1118_c0_g1_i1 |
870 |
674.936 |
4,759.73 |
58,974.33 |
|
TRINITY_DN594_c0_g1_i5 |
285 |
88.080 |
4,616.79 |
6,653.94 |
|
TRINITY_DN1537_c0_g1_i1 |
336 |
147.497 |
4,145.08 |
9,779.33 |
|
TRINITY_DN1537_c0_g2_i1 |
319 |
130.788 |
4,080.68 |
8,518.40 |
|
TRINITY_DN1537_c0_g1_i3 |
304 |
115.273 |
3,896.85 |
7,899.20 |
|
TRINITY_DN582_c1_g1_i3 |
189 |
35.613 |
3,852.78 |
2,893.85 |
Топ-10 транскриптов для reference_rep1:
|
Транскрипт |
Длина (bp) |
EffectiveLength |
TPM |
NumReads |
|---|---|---|---|---|
|
TRINITY_DN538_c0_g1_i1 |
211 |
51.106 |
13,152.02 |
13,610.73 |
|
TRINITY_DN510_c2_g3_i1 |
205 |
47.464 |
8,580.85 |
8,247.35 |
|
TRINITY_DN1458_c0_g1_i6 |
811 |
618.453 |
8,103.15 |
101,479.17 |
|
TRINITY_DN510_c1_g6_i1 |
225 |
60.069 |
7,996.41 |
9,726.61 |
|
TRINITY_DN538_c0_g2_i1 |
204 |
46.875 |
7,520.91 |
7,138.82 |
|
TRINITY_DN538_c0_g2_i2 |
351 |
165.883 |
6,742.01 |
16,412.98 |
|
TRINITY_DN510_c1_g1_i6 |
233 |
66.392 |
5,891.13 |
7,522.67 |
|
TRINITY_DN538_c0_g3_i1 |
419 |
229.360 |
5,879.14 |
16,312.52 |
|
TRINITY_DN510_c2_g3_i4 |
209 |
48.158 |
5,664.45 |
5,605.84 |
|
TRINITY_DN510_c1_g2_i2 |
437 |
247.783 |
5,550.68 |
13,680.38 |
Топ-10 транскриптов для reference_rep2:
|
Транскрипт |
Длина (bp) |
EffectiveLength |
TPM |
NumReads |
|---|---|---|---|---|
|
TRINITY_DN24_c0_g1_i26 |
855 |
659.384 |
8,305.99 |
106,518.13 |
|
TRINITY_DN865_c1_g1_i2 |
209 |
48.278 |
7,146.24 |
6,710.03 |
|
TRINITY_DN24_c0_g1_i6 |
647 |
451.414 |
6,772.98 |
59,463.23 |
|
TRINITY_DN6819_c0_g1_i1 |
323 |
135.638 |
6,679.26 |
17,619.89 |
|
TRINITY_DN478_c0_g1_i5 |
329 |
141.005 |
6,238.81 |
17,109.25 |
|
TRINITY_DN865_c1_g2_i2 |
275 |
95.417 |
6,136.98 |
10,955.65 |
|
TRINITY_DN865_c1_g3_i3 |
416 |
226.457 |
5,789.21 |
16,029.12 |
|
TRINITY_DN24_c0_g1_i2 |
348 |
157.516 |
5,607.67 |
12,826.78 |
|
TRINITY_DN6819_c0_g2_i1 |
489 |
298.245 |
5,356.89 |
25,172.13 |
|
TRINITY_DN478_c0_g1_i6 |
488 |
297.631 |
5,320.14 |
25,077.61 |
Топ-10 транскриптов для reference_rep3:
|
Транскрипт |
Длина (bp) |
EffectiveLength |
TPM |
NumReads |
|---|---|---|---|---|
|
TRINITY_DN470_c1_g3_i1 |
201 |
44.245 |
9,357.93 |
7,518.68 |
|
TRINITY_DN222_c0_g1_i9 |
248 |
74.530 |
8,910.76 |
12,059.98 |
|
TRINITY_DN1233_c0_g1_i10 |
624 |
427.358 |
6,816.02 |
52,895.81 |
|
TRINITY_DN11156_c1_g1_i1 |
281 |
99.761 |
5,067.67 |
9,180.52 |
|
TRINITY_DN1233_c0_g1_i34 |
769 |
550.311 |
4,617.63 |
46,145.14 |
|
TRINITY_DN11156_c1_g1_i5 |
365 |
175.837 |
4,493.25 |
13,284.88 |
|
TRINITY_DN470_c1_g1_i2 |
431 |
241.768 |
4,482.77 |
12,437.33 |
|
TRINITY_DN222_c0_g1_i4 |
337 |
146.527 |
4,343.44 |
8,836.09 |
|
TRINITY_DN11156_c1_g2_i1 |
254 |
80.682 |
4,194.82 |
6,767.08 |
|
TRINITY_DN470_c1_g2_i1 |
247 |
73.864 |
4,041.87 |
5,424.80 |


