Изучение антибиотикорезистентности и молекулярное типирование нозокомиальных штаммов Klebsiella pneumoniae, циркулирующих в стационарах г. Нижнего Новгорода
Конференция: XI Международная научно-практическая конференция «Научный форум: медицина, биология и химия»
Секция: Микробиология

XI Международная научно-практическая конференция «Научный форум: медицина, биология и химия»
Изучение антибиотикорезистентности и молекулярное типирование нозокомиальных штаммов Klebsiella pneumoniae, циркулирующих в стационарах г. Нижнего Новгорода
STUDY OF ANTIBIOTIC RESISTANCE AND MOLECULAR TYPING OF KLEBSIELLA PNEUMONIAE NOSOCOMIAL STRAINS CIRCULATING IN NIZHNY NOVGOROD HOSPITALS
Dmitriy Gorev
graduate student, National Research N.I. Lobachevsky State University of Nizhny Novgorod (UNN), Russia, Nizhny Novgorod
Anna Tochilina
candidate of Biological Science, Blokhina Scientific Research Institute of Epidemiology and Microbiology of Nizhny Novgorod, Russia, Nizhny Novgorod
Alexander Rechkin
candidate of Biological Sciences, assistant professor, National Research N.I. Lobachevsky State University of Nizhny Novgorod (UNN) Russia, Nizhny Novgorod
Irina Belova
candidate of Medical Sciences, Blokhina Scientific Research Institute of Epidemiology and Microbiology of Nizhny Novgorod, Russia, Nizhny Novgorod
Irina Solovyeva
doctor of Biological Sciences, Blokhina Scientific Research Institute of Epidemiology and Microbiology of Nizhny Novgorod, Russia, Nizhny Novgorod
Аннотация. В статье представлены результаты изучения резистома и молекулярного типирования 63 нозокомиальных штаммов K. pneumoniae.
Все штаммы были идентифицированы с использованием MALDI TOF масс-спектрометрии, установлено, что все штаммы относились к виду Klebsiella pneumoniae ssp. pneumoniae. При изучении резистома выявлено, что более половины исследуемых штаммов (52,4 %) не имели генетических маркеров устойчивости, 28,6 % изолятов обладали сочетанной антибиотикорезистентностью и несли детерминанты β-лактамаз CTX-M и OXA-48, 19 % изолятов обладали одной из идентифицированных детерминант.
На последнем этапе работы было проведено генотипирование исследуемой выборки штаммов с использованием метода RAPD. В результате анализа электрофоретических паттернов анализируемые изоляты были объединены в четыре независимых кластера. Была отмечена кластеризация анализируемых штаммов в соответствии с местом их выделения.
Abstract. The article presents the results of study of resistance and molecular typing of 63 K. Pneumoniae nosocomial strains. All strains were identified using MALDI TOF mass-spectometry, which confirmed that all strains belonged to the Klebsiella pneumoniae ssp. pneumoniae species. The resistance study revealed that over the half of the studied strains (52.4 %) had no genetic resistance markers; 28.6 % of isolates had a complex antibiotic resistance and determinants of СТХ-М and ОХА-48 β- lactomases; 19 % of isolates had one of the identified determinants. On the last stage, the investigated strain samples underwent genetic typing using the RAPD method. It helped to combine the analyzed isolates into four independent clusters according to their relevant electrophoretic patterns. It also distinguished clustering of analyzed strains in accordance with their identification location.
Ключевые слова: K. pneumoniae; нозокомиальная инфекция; устойчивость к антибиотикам; Масс-спектрометрия MALDI TOF; RAPD.
Keywords: K. pneumoniae, nosocomial infection, antibiotic resistance, MALDI TOF mass spectrometry, RAPD.
Одной из наиболее серьезных проблем в эпидемиологии являются нозокомиальные (внутрибольничные) инфекции. Широко распространенным возбудителем внутрибольничных инфекций в стационарах России в настоящее время выступает Klebsiella pneumoniae [2]. К группе риска по заболеваемости относятся пациенты перенесшие хирургические вмешательства, пациенты отделений ОРИТ, неонатологии и неонатальной реанимации [6, 7].
По данным научной литературы 45,2 % штаммов Klebsiella pneumoniae являются продуцентами различных детерминант антибиотикорезистентности, в том числе β-лактамаз и карбапенемаз - ферментов, разрушающих антибиотики посредством гидролитического расщепления циклической амидной связи в их структуре [5]. Число микроорганизмов устойчивых к действию современных антибиотиков среди представителей этого вида растет с каждым годом, ставя под удар эффективность антибактериальной терапии.
Ввиду сложившейся ситуации возникает необходимость исследования нозокомиальных штаммов Klebsiella pneumoniae на наличие детерминант антибиотикорезистентности и их эпидемиологического типирования.
Для точной видовой идентификации на данный момент целесообразно использовать MALDI-TOF масс-спектрометрию. Несомненные преимущества метода: простота исполнения, воспроизводимость и экономическая выгодность [1, 11].
В настоящее время широко распространены молекулярно-генетические методы детекции детерминант антибиотикорезистентности с использованием ПЦР, в том числе и ПЦР в реальном времени [10]. Для типирования микроорганизмов используют метод мультилокусного сиквенс-типирования (MLST), электрофорез в пульсирующем поле (PFGE), метод анализа полиморфизма длины рестрикционных фрагментов (RFLP), метод анализа полиморфизма амплифицированных фрагментов ДНК (RAPD) [3].
Необходимо отметить, что метод RAPD обладает высокой чувствительность и позволяет дифференцировать близкородственные виды, прост в исполнении и экономически выгоден.
Целью данной работы явилась точная видовая идентификация, изучение резистома и типирование нозокомиальных штаммов K. pneumoniae, циркулирующих в стационарах г. Н. Новгорода, с использованием методов MALDI TOF масс-спектрометрии и метода RAPD.
Материалы и методы исследования
На первом этапе проводили идентификацию 63 нозокомиальных штаммов K. pneumoniae из двух стационаров города Н. Новгорода с использованием MALDI TOF масс-спектрометра Autoflex (Bruker, Германия) и программно-аппаратного комплекса Biotyper 3.0 (Bruker, Германия). Для этого выращивали 24-часовую культуру микроорганизмов на мясо-пептонном агаре и проводили идентификацию согласно инструкции производителя. О достоверности идентификации судили по значению коэффициента совпадения (Score values) - 2,000 – 3,000 – идентификация до вида, 1,999-1,700 – идентификация до рода, 1,699 – 0 – идентификация не прошла) и значению категорий – А – достоверная идентификация до вида, В - достоверная идентификация до рода, С – недостоверный результат.
Далее проводили детекцию генов резистентности к антибиотикам. Для экстракции ДНК использовали коммерческий набор «ДНК-экспресс», фирмы «Литех», Москва. Обнаружение генов BLATEM и BLASHV производили с использованием специфических олигонуклеотидных праймеров [9, 10], а детерминант BLAOXA-48, BLACTX-M, BLAIMP, BLAKPC, BLAVIM, BLANDM - с использованием коммерческих тест-систем фирмы «Литех», Москва.
На последнем этапе проводили RAPD с использованием протокола, представленного в научной литературе [12]. Для анализа полученных профилей RAPD использовали интернет-ресурс http://insilico.ehu.es/dice_upgma [8].
Результаты и их обсуждение
В ходе проведения исследования с использованием MALDI TOF масс-спектрометрии все штаммы были идентифицированы как Klebsiella pneumoniae ssp. pneumoniae, значения Score values варьировали от 2,25 до 2,4.
В результате идентификации генетических детерминант резистентности к антибиотикам у исследуемых штаммов было установлено, что более половины анализируемых штаммов (52,4 %) не имеют генетических маркеров устойчивости, 28,6 % изолятов несут детерминанты BLACTX-M и BLAOXA-48., то есть обладают сочетанной резистентностью, 19 % изолятов обладают лишь одной из названных выше детерминант. Наличие генов β-лактамаз BLATEM и BLASHV и карбапенемаз BLAIMP, BLAKPC, BLAVIM, BLANDM выявлено не было. Необходимо отметить различия резистомов штаммов в зависимости от места выделения. А первом стационаре циркулируют штаммы, не несущие детерминант резистентности, а во втором – практически все обладают либо сочетанной резистентностью, либо несут одну детерминанту.
B-лактамаза CTX-M относится к сериновым бета-лактамазам класса А и обуславливает устойчивость к пенициллинам и цефалоспоринам, причем среди ферментов этого типа часто встречаются ферменты расширенного спектра (БЛРС). OXA-48 относится к сериновым бета-лактамазам класса D, с высокой карбапенемазной активностью и может обуславливать устойчивость к карбапенемам.
Поскольку обнаруженные детерминанты сопряжены с высоко трансмиссивными мобильными генетическими элементами (CTX-M) или имеют плазмидную локализацию (OXA-48), это способствует их распространению. Причем штаммы, несущие комплекс детерминант по данным научной литературы опасны в плане формирования панрезистентности, что может привести к возникновению высоко резистентного госпитального штамма, устойчивого к пенициллинам, цефалоспоринам и карбапенемам. В этом случае встает необходимость привлечения антибиотиков резерва [4].
Генотипирование с использованием RAPD анализа позволило получить индивидуальную электрофоретическую картину (паттерн) для каждого исследуемого штамма и объединить их в четыре кластера (рис.1).
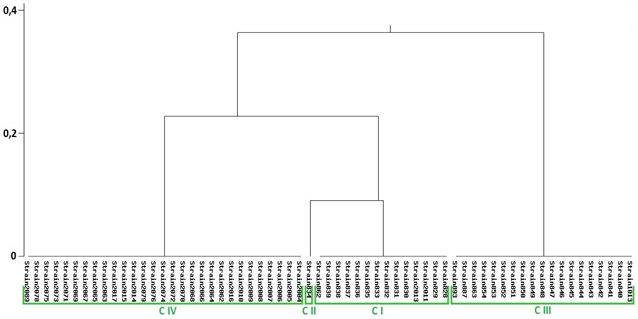
Рисунок 1. RAPD-дендрограмма нозокомиальных штаммов K. pneumoniae, циркулирующих
в стационарах г. Н. Новгорода
Выявлено, что каждый RAPD-кластер объединяет штаммы со сходным резистомом: кластеры CI и CII формируют штаммы не несущие детерминант устойчивости; кластеры CIII и CIV представлены изолятами несущими одну/две детерминанты устойчивости, с преобладанием в четвертом кластере штаммов, обладающих сочетанной устойчивостью (BLACTX-M и BLAOXA-48.) (табл. 1).
Таблица 1.
Кластеризация штаммов K. pneumoniae на основе электрофоретических паттернов RAPD-типирования
|
№ |
Кластер CI |
кластер CII |
кластер CIII |
кластер CIV |
|
Номера штаммов |
828, 829, 830, 831, 832, 833, 835, 836, 837, 838, 839, 862, 2011, 2013 |
834 |
840, 841, 842, 843, 844, 845, 846, 847, 848, 849, 850, 851, 852, 853, 854, 863, 887, 893, 1013 |
2004, 2005, 2006, 2007, 2008, 2009, 2010, 2014, 2015, 2016, 2017, 2062, 2063, 2064, 2065, 2066, 2067, 2068, 2069, 2070, 2071, 2072, 2073, 2074, 2075, 2076, 2078, 2079, 2089 |
|
Выявленные генетические детерминанты антибиотикорезистентности |
Нет
|
Нет |
BLACTX-M BLAOXA-48 |
BLACTX-M BLAOXA-48 |
Отмечалась зависимость кластеризации и от места выделения штаммов, то есть штаммы из одного стационара объединялись в единый кластер.
Таким образом, метод RAPD целесообразно использовать как один из компонентов молекулярно-эпидемиологического мониторинга в системе эпидемиологического надзора за штаммами Klebsiella pneumoniae потенциальными возбудителями нозокомиальных инфекций. Метод обладает достаточной разрешающей способностью, прост в исполнении и экономически выгоден.
Заключение
В ходе данной работы с использованием MALDI TOF масс-спектрометрии было установлено точное таксономическое положение 63 нозокомиальных штаммов клебсиелл, все штаммы идентифицированы как Klebsiella pneumoniae ssp. pneumoniae.
В результате исследования на наличие детерминант устойчивости к антибиотикам было установлено, что более половины исследуемых штаммов (52,4 %) не имеют генетических маркеров устойчивости, 28,6 % изолятов обладают сочетанной устойчивостью и содержат гены β-лактамаз CTX-M и OXA-48, 19 % изолятов обладают одной из вышеназванных детерминант.
Генотипирование исследуемой выборки штаммов с использованием метода RAPD позволило объединить анализируемые изоляты в четыре независимых кластера, в соответствии с присущими штаммам электрофоретическими паттернами. Была отмечена кластеризация анализируемых штаммов в соответствии с местом их выделения и характером резистома.
Циркуляция в стационарах города штаммов Klebsiella pneumoniae, обладающих сочетанной устойчивостью, которая обусловлена β‑лактамазой BLACTX-M и карбапенемазой BLAOXA-48, может способствовать распространению этих детерминант вследствие горизонтального переноса и является фактором риска по формированию клинически опасных панрезистентных штаммов энтеробактерий. Это подтверждает необходимость молекулярно-генетического мониторинга с целью прогнозирования эпидемической ситуации и своевременного вмешательства в ход эпидемического процесса.


